2022年6月8日,国际学术期刊《PLOS Computational Biology》在线发表了题为《Identification of upstream transcription factor binding sites in orthologous genes using mixed Student’s t-test statistics》(“使用混合t检验统计识别同源基因中的转录因子结合位点”)的研究论文。报道了我院黄廷华副教授团队(http://www.thua45.cn)在转录因子结合位点预测工具开发方面的研究成果。
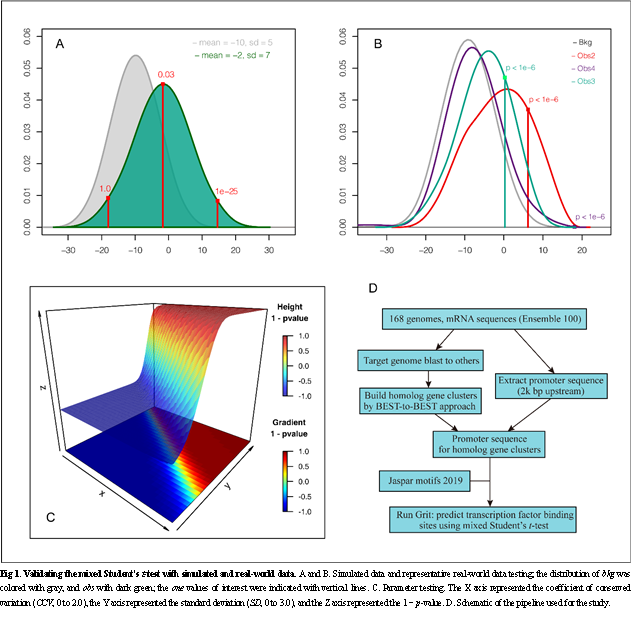
转录因子通过与启动子序列Motif结合来调节DNA到信使RNA的转录。识别全基因组中Motif的位置需要大量计算。定位基因组中的转录因子结合位点并确定其功能是解读各种生物过程的基础。另外,准确的转录因子结合位点预测可以节省传统实验的成本和时间,全基因组的转录因子结合位点预测还可以从系统生物学的角度为转录组的调节研究提供新的见解。
该研究开发了一个名为“Grit”的计算工具,通过将转录因子与具有同源性的基因的启动子序列相匹配来筛选转录因子结合位点。该工具采用了一种新开发的混合t检验统计方法,利用物种间的保守信息检测高分结合位点。该程序在四核Amazon服务器上以3.2 Mbp/s的速率执行序列扫描,并已通过Chip-Seq数据集的基准测试,Grit是目前性能最好的转录因子结合位点预测工具之一。在ChIP-Atlas数据集的校准测试中,它显著优于著名的转录因子结合位点预测工具Pscan(4.8%)和FIMO(17.8%)。Grit可通过主页http://www.thua45.cn/grit或Git-hub以Academic-free许可证的方式免费获得。
长江大学我院黄廷华副教授为该论文的第一作者,该团队的研究生肖红为第二作者,此外研究生田棋、何珍参与了部分工作。长江大学2018届大创项目成员杨敏(主持人)、刘金徽、董凯辉、徐明江、陈智、祝世家,2019届大创项目成员蒋彩云(主持人)、李勇霞、李承隆、何亮、江山,2020届大创项目成员林泽钊(主持人)、袁程等为本项目的数据收集做出了重要贡献。长江大学我院高学军教授、姚敏副教授为论文的通讯作者。
